|
Edler Genuss
Edler GenussDas Chloroplastengenom im Fokus der Authentizitätsüberprüfung von Lebensmitteln am Beispiel des ecuadorianischen EdelkakaosSchokolade und andere Kakaoprodukte waren schon immer beliebte Lebensmittel, erinnern an Genuss und werden mit einem positiven Lebensgefühl verbunden. Denkt man an das Produktdesign, die Verpackungsgestaltung und auch an den Preis z.?B. einiger Bitterschokoladen, so stellt man fest, dass es sich um edle Produkte handeln muss, wie es bei anderen Lebensmitteln wie bspw. Wein oder Kaffee der Fall ist. Dementsprechend findet der Verbraucher auf Kakaoprodukten als Qualitätsmerkmal auch immer öfter Angaben zum Ursprungsort der verarbeiteten Kakaobohnen. Die Nachfrage nach derlei Lebensmitteln durch den Verbraucher ist stetig steigend, sodass auch die Produktion von Edelkakaosorten (aktuell etwa 5?% der gesamten Kakaoproduktion) damit einhergehend zunehmen muss. Edelkakaoprodukte erfreuen sich immer größerer Beliebtheit Arriba ist die wohl wichtigste Edelkakaosorte. Sie macht etwa die Hälfte des produzierten Edelkakaos aus, wird vorwiegend in Ecuador angebaut und ist für ihr einzigartiges und sehr intensives Aroma bekannt. Neben dem Arribakakao wird in Ecuador seit den 70er-Jahren eine weitere Sorte angebaut [1, 2]. Es handelt sich um den Konsumkakao CCN 51 – ein Klon, der zwar ertragreicher, robuster und resistenter ist, aber im Gegensatz zum anfälligeren Arriba ein schwächeres Aroma aufweist [3, 4]. Da die Kultivierung des CCN 51 ein geringeres Ernterisiko bedeutet, wird diese Sorte von den ecuadorianischen Kakaobauern vorzugsweise angebaut. Eine zunehmende Produktion des Konsumkakaos steht allerdings im Gegensatz zum wachsenden Bedarf an Edelkakao des Konsumenten. Es liegt nahe zu vermuten, dass die beiden ecuadorianischen Kakaosorten miteinander vermischt werden, woraus eine verminderte Qualität der entstehenden Produkte resultiert. Eine Vermengung könnte absichtlich erfolgen, um durch den Verkauf eines vermeintlich reinen Edelkakaos höhere Einnahmen zu erzielen. Aber auch auf ungewolltem Wege könnte der kostengünstigere Kakao in die Chargen des edleren gelangen, da beide Sorten oftmals nebeneinander angebaut und verarbeitet werden. Aus genannten Gründen besteht ein reges Interesse an einer Methode, mit der es möglich ist, die beiden Kakaosorten voneinander zu unterscheiden (Abb. 1).
DNA – ein geeigneter Biomarker Bei allen in einer pflanzlichen Zelle zur Verfügung stehenden Biomarkern zeichnet sich die DNA bspw. gegenüber Proteinen und Stoffwechselprodukten dahingehend aus, dass sie für jeden Organismus individuell ist. Im Weiteren ist sie konstant, also im Rahmen der für die Lebensmittelanalytik entscheidenden Zeitspanne unabhängig von äußeren Einflüssen. Neben dem Kerngenom verfügt jede pflanzliche Zelle über ein Chloroplastengenom (= plastidäre DNA). Da es sich bei CCN 51 und Arriba um sehr stark miteinander verwandte Organismen handelt, ist die Suche nach Sequenzunterschieden in der plastidären DNA eher geeignet als in der nukleären, da von Chloroplastengenomen bekannt ist, dass sie im Vergleich zum Kerngenom eine höhere Variabilität aufweisen [5]. Am Anfang müssen umfangreiche Sequenzierungsarbeiten durchgeführt werden. Üblicherweise sollte für eine Sequenzierung des gesamten Chloroplastengenoms dieses isoliert vom Kerngenom vorliegen. Entsprechend KANE et al. (2012) entfallen aufwändige Trennungsschritte der beiden Genome erfreulicherweise: Pro Zelle kommt die nukleäre DNA in einfacher Kopie vor. Dieselbe Pflanzenzelle verfügt über etwa drei bis zehn Chloroplasten, die jeweils etwa 70 Kopien ihrer DNA besitzen. Auch wenn das Chloroplastengenom etwa 2000-mal kleiner ist als das Kerngenom, kommt es pro Zelle wesentlich häufiger vor. Diese Tatsache bildet die Grundlage für die von KANE et al. entwickelte Methode „Low coverage whole genome shotgun sequencing“: Aufgrund der geringen Abdeckung während der Sequenzierung werden die „high copy“-Fraktionen erfasst. Auch in Anwesenheit der nukleären DNA kann dadurch die plastidäre DNA sequenziert werden [6].
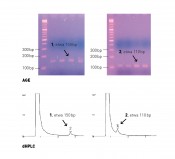
Abb.2 Detektion nach der PCR-RFLP mittels AGE und CGE. Nach einer PCR werden PCR-Produkte erhalten (1 und 2, Vor Restriktion, AGE). Das Amplifikat 1 enthält die Erkennungssequenz des eingesetzten Restriktionsenzyms und wird verdaut: Es werden zwei Fragmente bei etwa 170bp und 260bp detektiert (1, nach der Restriktion, AGE und CGE). Amplifikat 2 enthält aufgrund eines SNPs nicht die Erkennungssequenz und wird nicht verdaut: Anstelle von zwei Fragmenten wird auch nach der Restriktion das unfragmentierte PCR-Produkt bei etwa 420bp detektiert (2, nach der Restriktion, AGE und CGE). BW = Blindwert. SNPs im Chloroplastengenom Ein Vergleich der Chloroplastengenome der beiden Kakaosorten CCN 51 und Arriba zeigt wie erwartet lediglich minimale Abweichungen (etwa 0,03%). Es handelt sich hierbei lediglich um Einzelbasenaustausche (SNPs = Single Nucleotide Polymorphisms), die eine Anwendung einer einfachen PCR (= Polymerase Chain Reaction) erschweren. Da sich jedoch einige wenige von diesen in der Erkennungssequenz eines Restriktionsenzyms befinden, können diese SNPs sogar für eine Kakaosortendifferenzierung genutzt werden [7]. Zunächst wird eine sortenunspezifische PCR durchgeführt, bei der die Primer den SNP weiträumig flankieren. Für beide Kakaosorten wird also ein PCR-Produkt erhalten, das in einem anschließenden Schritt – der RFLP (=Restriction Fragment Length Polymorphism) – mit einem Restriktionsenzym verdaut wird. Da diese Endonukleasen hochspezifisch sind, können sie nur das PCR-Produkt spalten, das die definierte Sequenz enthält. In einem letzten Schritt erfolgt die Detektion mittels AGE (=Agarosegelelektrophorese), CGE (=Kapillargelelektrophorese) oder dHPLC (=denaturierende HPLC). Im Falle eines erfolgten Verdaus aufgrund der korrekten Erkennungssequenz sind zwei Fragmente geringerer Größe zu beobachten. Enthält das Amplifikat nicht die genaue Sequenz, so bleibt ein Verdau aus und es wird anstelle von zwei Fragmenten das unverdaute PCR-Produkt detektiert. Entsprechend der Fragestellung des Projekts AiF/FEI 16796 N in Bezug auf eine Vermischung des ecuadorianischen Edelkakaos Arriba mit dem kostengünstigeren Konsumkakao CCN 51 kann die PCR-RFLP dahingehend entwickelt werden, dass im letzten Schritt nach dem enzymatischen Verdau die Sorte CCN 51 detektiert wird. Das heißt, dass im Falle der Anwesenheit von CCN?51 zwei Fragmente erhalten werden, da in der Sequenz des Konsumkakaos die korrekte Erkennungssequenz des Restriktionsenzyms enthalten ist (Abb. 2). Eine weitere Abweichung in der Sequenz des Chloroplastengenoms zeigte sich in der IRR (=Inverted Repeat Region), nachdem sie nach DHINGRA et al. (2005) mithilfe von 27 Primerpaaren partiell sequenziert worden ist [8]. Die IRR ist eine Sequenz, die entgegengesetzt komplementär zu einer im selben Genom liegenden Sequenz ist. Diese beinhaltet im vorliegenden Chloroplastengenom die fünfbasige Sequenz (TAAAG)n (Repeat), die in den zwei Sorten unterschiedlich oft wiederholt wird (Abb. 3). Da es sich hierbei nicht um qualitative Sequenzunterschiede handelt, sondern lediglich die Anzahl der Abfolge des Repeats in den beiden Kakaosorten verschieden ist, kann auch in diesem Falle keine PCR mit sortenspezifischen Primern entwickelt werden. Die unterschiedliche Anzahl der Wiederholung kann wiederum auch für eine Nachweismethode genutzt werden: Für beide Kakaosorten werden PCR-Produkte generiert, wobei die PCR-Region die IRR weiträumig flankiert. In einem darauffolgenden Schritt werden die erhaltenen PCR-Produkte mit AGE, CGE oder dHPLC detektiert. Es können daraufhin sortenabhängig Amplifikate beobachtet werden, die sich um 40bp voneinander unterscheiden, da die fünf Basen lange Sequenz in einer Sorte 6-mal (TAAAG)6 und in der anderen 14-mal (TAAAG)14 wiederholt wird (Abb. 4).
Abb.3 Ausschnitt des Alignments nach der Sequenzierung der IRR. Das Repeat (TAAAG)n kommt in einer Kakaosorte 6-mal und in der anderen 14-mal vor. Alternative Methoden Die Sequenzunterschiede zwischen CCN?51 und Arriba beschränken sich auf SNPs oder eine unterschiedliche Repeat-Wiederholung im Chloroplastengenom. Es konnten – basierend auf diesen Abweichungen – PCR-Methoden entwickelt werden, mit denen eine Vermischung des Edelkakaos mit dem Konsumkakao nachgewiesen werden kann. Alternativ könnten auch andere Methoden auf Grundlage der genannten Sequenzunterschiede entwickelt werden. Ist ein SNP vorhanden, kann die LPA (=Ligation-Dependent Probe Amplification) genutzt werden. Zwei Primer-Sonden hybridisieren in direkter Nachbarschaft zueinander an eine Target-Sequenz. Im nächsten Schritt erfolgt die kovalente Verbindung dieser beiden Sorten durch eine hochspezifische Ligase. Eine Ligation kann allerdings nur dann erfolgen, wenn die Hybridisierung an die Target-Sequenz lückenlos erfolgte, was im Falle eines SNPs nicht der Fall ist. Es folgt die PCR, wobei die notwendigen Primer bereits an die Sonden hybridisiert sind. Die Entstehung eines PCR-Produkts ist jedoch nur im Falle von ligierten Sonden gegeben. Ist ein SNP anwesend, bleiben die Ligation und somit auch die PCR aus. Es kann kein PCR-Produkt nachgewiesen werden [9]. Über den Vergleich von Mikrosatelliten könnten ebenfalls Arbeiten zur Differenzierung stattfinden. Mikrosatelliten sind kurze und nicht kodierende Genom-Sequenzen, die sich am selben Locus wiederholen, wie bereits im Falle der IRR im Kakaochloroplastengenom beschrieben worden ist. Die Wiederholungen treten in der Regel in unterschiedlicher Häufigkeit auf. Auch in der nukleären DNA kommen Mikrosatelliten vor [10, 11, 12]: Nach der Analyse und Detektion der aus CCN 51 und Arriba erhaltenen PCR-Produkte, die entsprechend der Publikationen aus dem Kerngenom erhalten werden, könnten auch so entsprechende Rückschlüsse auf die Sorten erhalten werden.
Abb.4 Detektion des PCR-Produkts nach einer sortenunspezifischen PCR, bei der die Primer das (TAAAG)n-Repeat flankieren. Da das Repeat in den Sorten (1:(TAAAG)14 und 2:(TAAAG)6) unterschiedlich häufig wiederholt wird, ergeben sich PCR-Produkte, die sich um 40 bp voneinander unterscheiden (1: etwa 150 bp und 2:etwa 110 bp). Fazit Authentizitätsüberprüfungen von pflanzlichen Lebensmitteln stellen eine interessante Herausforderung der Lebensmittelanalytik dar. Es ist mit weiteren Innovationen und Technologien im Bereich der Sequenzierungen zu rechnen. Da sie zudem schneller und weniger kostenintensiv sind als vor etlichen Jahren, ist es möglich, komplette Genome in kürzester Zeit zu entschlüsseln. Damit einhergehend ist eine rasante Methodenentwicklung zu verzeichnen, mit deren Hilfe die Problemstellung der Authentizität von Lebensmitteln eine neue Dimension erreicht. Die skizzierte Durchführung anhand des Kakaos ist beispielhaft. Es ist anzunehmen, dass eine ähnliche Vorgehensweise auf eine Vielzahl anderer Rohstoffe und somit auch auf Lebensmittel übertragbar ist.
Literatur Foto: © panthermedia.net|Viet Doan |
L&M 3 / 2014
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Die Autoren:Weitere Artikel online lesenNewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |