|
NcRNAs - Physiologische Prozesse
NcRNAs - Physiologische ProzesseVersteckte Dirigenten
Prof. Dr. Alexander Hüttenhofer und Dr. Mathieu Rederstorff, Institut für Genomik und RNomik, Medizinische Universität Innsbruck, Biozentrum
Man nimmt daher an, dass ein signifikanter Anteil der restlichen 88,5 % von RNA-Transkripts als Quelle für regulatorische ncRNA dient. Diese Befunde setzen daher eine bisher verborgene Ebene regulatorischer Elemente im menschlichen und anderen eukaryotischen Genomen voraus. Vermutlich sind bis zu 450.000 ncRNAGene im menschlichen Genom kodiert. Viele ncRNAs (Tab. 1), z.B. ribosomale RNAs (rRNAs) oder mikro-RNAs (miRNAs) (Abb. 2) spielen eine wichtige Rolle bei physiologischen Prozessen und vermutlich auch bei der Entstehung von Erkrankungen, einschließlich Krebs. ncRNAs in pathogenen Organismen Bakterien und Viren nutzen die Plastizität und Eigenschaften von ncRNAs, um Pathogenität zu entwickeln und die Invasion in den Wirtsorganismus vorzubereiten. Erstmals wurden aus der Klasse der ncRNAs sogenannte miRNAs (mi = micro) im Genom von DNA-Viren der Herpesfamilie beschrieben, kurz nachdem man diese ncRNAs bei Säugetieren [2] entdeckt hatte. Über 160 virale miRNAs sind inzwischen bekannt und neue werden ständig entdeckt. Kürzlich wurden zwei neue Epstein-Barr-Virus-miRNAs (EBV) in Gewebeproben eines Nasopharynxkarzinoms identifiziert, die nie zuvor in einem infizierten Gewebe beobachtet wurden. Außerdem fand man drei neue miRNAs, die nur während der Latenzphase des Herpes-Simplex Virus (HSV) 2 exprimiert werden [3].
Tab. 1 Prinzipielle metazoische, zelluläre RNAs.
Abb. 1 Vereinfachtes Schema der miRNA-Biogenese und Funktion in Metazoa MiRNAs werden im Zellkern durch die RNA-Polymerase II als miRNA-Vorläufer (pri-miRNAs) transkribiert, welche durch die RNase III gespalten werden, ein Enzym der Drosha Familie (in violet). Es entstehen pre-miRNAs mit charakteristischer Haarnadelstruktur ("hairpin"). Diese werden mithilfe von Exportin 5 (in gelb) in das Zytoplasma exportiert und dort durch die RNase Dicer (in rot) in 19-25nt lange RNA-Duplece zerteilt. Ein Strang der Duplexe, die reife miRNA, assoziiert nun mit dem RNA-induced silencing complex (RISC, in grün). Die miRNA führt den RISC Komplex zu der 3'-untranslated Region (3'-UTR) der Ziel-mRNA, hieraus resultiert die translationale Repression der Ziel-mRNA. Rederstorff M. und Hüttendorfer A., (2010), Current Opinion in Molecular Therapeutics.
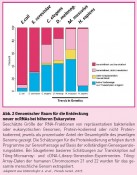
Abb. 2 Genomischer Raum für die Entdeckung neuer ncRNAs bei höheren Eukaryonten Geschätzte Größe der RNA-Fraktionen von repräsentativen bakteriellen oder eukaryotischen Genomen, Protein-kodierend oder nicht Protein-kodierend, jeweils als prozentualer Anteil der Gesamtgröße des jeweiligen Genoms gezeigt. Die Schätzungen für die Proteinkodierung erfolgen durch Programme zur Genvorhersage auf Basis der vollständigen Gensequenzierungsdaten. Bei Säugetieren basieren Schätzungen zur Transkription auf Tiling-Microarray- und cDNA-Library-Generation-Experimenten. Tiling-Array-Daten der humanen Chromosomen 21 und 22 für das gesamte menschliche Genom extrapoliert. Adaptiert aus Hüttenhofer A. et al., Trends. Genet. 2005
Mit einigen Ausnahmen ist die Funktion der meisten miRNA bisher unbekannt. Von miRNAs des Karposisarkom-assoziierten Herpesvirus (KHSV) weiß man, dass sie direkt in die Pathogenese involviert sind, indem sie direkt auf die Thrombospondin1 (THBS1) mRNA einwirken. Diese kodieren für ein Wirtsprotein, das die Zelladhäsion und -migration reguliert [4]. MiR-UL112, das auf dem Genom des humanen Cytomegalievirus (HCMV) reprimiert, zielt auf die humane Major Histocompatibility Complex Klasse I B mRNA (MICB), eine Komponente der natürlichen Killerzellen, deren Expression normalerweise bei viralen Infekten hochreguliert wird [5]. Als Konsequenz wird die Verbreitung des Virus durch eine verringerte Aktivität der natürlichen Killerzellen begünstigt. miR-UL 12 interagiert aber auch mit verschiedenen viralen mRNAs. Diese kodieren für in die virale Replikation eingebundenen Proteine und reduzieren damit die Zahl der Virus-Kopien. Ähnlich exprimiert der Simian-Virus 40 (SV-40) miRNAs. Sie zielen auf die viralen mRNAs, welche T-Zell-Antigene kodieren, um der zytotoxischen T-Zell-Antwort des Wirts zu entgehen [6]. Das HSV-1 Genom exprimiert zwei miRNAs, welche die virale Latenz in Neuronen fördern durch Interaktion mit den ICPO und ICP4 mRNAs. Diese kodieren zwei für die Replikation und Infektion erforderliche virale Proteine [7]. Viren können also die Immunantwort des Wirtsorganismus entweder durch Reduktion der Antigensynthese oder durch eine direkte Schwächung der angeborenen Immunantwort des Wirtes modulieren. Viren nutzen auch längere ncRNAs, um die Immunantwort des Wirtsorganismus zu schwächen. Adenoviren kodieren zwei 165 Nukleotide lange, virusassoziierte RNAs (VA RNAs), welche die antivirale Proteinkinase R (PKR) blockieren. VA RNAs lassen sich in eine konservierte dsRNA-Struktur falten und akkumulieren bis zu 108 Kopien pro Zelle, wodurch sie Exportin 5 für ihren Export in das Zytoplasma anreichern (Abb. 2) [8]. Außerdem besitzen VA RNAs und pre-miRNAs ähnliche Strukturen. VA RNAs sind in der Lage, die Ribonuklease Dicer zu blockieren [8]. Ein geringer Anteil der VA RNAs kann trotzdem in 3 adenovirale miRNAs umgewandelt werden [8]. Zumindest ein wichtiger zellulärer Zielort konnte für mivaRNAI-138 identifiziert werden, nämlich der Splicing- und Translationsregulator TIA-1 mRNA [8]. Das Herpesvirus Saimiri kodiert sieben Herpesvirus-spezifische U RNAs (HSURs), während EBV die beiden ncRNAs (EBER) 1 und 2 mit einer Größe von etwa 170 nt kodiert. Ursprünglich wurde angenommen, dass EBER 1 und 2 nur im Zellkern lokalisiert sind. Es zeigte sich aber, dass EBER 1 und 2 von infizierten B Zellen freigesetzt werden und dabei eine angeborene Immunität durch Signale des Toll-like-Rezeptors 3 (TLR3) aktivieren [9]. Zusätzlich zu den 25 miRNAs und EBER 1 und 2-ncRNAs wurde nachgewiesen, dass das Genom von EBV die erstmals beschriebene, virale snoRNA – v-snoRNA1 [10] – kodiert. Obwohl ihre Funktion noch nicht völlig geklärt ist, wurde gezeigt, dass sie zu einer 24 nt langen miRNA-artigen Spezies prozessiert wird [10]. Ein mögliches Ziel für diese „sno-derived“ miRNA wurde in der 3´-UTR der viralen DNAPolymerase BALF5 mRNA lokalisiert. Von dieser nimmt man an, dass sie das Ziel einer anderen EBV-kodierten miRNA, miR-BART2 ist [2,10]. Einige Viren nutzen sogar die zelluläre Maschinerie oder ncRNAs des Wirts für ihre eigenen Zwecke. So kann sich HIV-1 nur replizieren, indem es die Wirts-tRNA3lys als Primer für die reverse Transkription seiner genomischen RNA nutzt, denn tRNA3lys ist als Primer komplementär zur primären Bindungsstelle (PBS) auf der viralen RNA. Die Wirtszellen von Säugetieren nutzen ebenfalls auf miRNA basierende Mechanismen, um sich gegen virale Infektionen zu wehren. MiR-29a ist bei HIV-1 infizierten T-Lymphozyten überexprimiert und zielt auf die 3'-UTR des viralen RNA-Genoms und unterbindet damit die Replikation des Virus [11]. Auch zelluläre miRNAs verhindern die Amplifikation des Retrovirus PFV-1 in menschlichen Zellen [12]. Zelluläre miRNAs scheinen also über eine zufällige Erkennung der viralen mRNAs neue antivirale Eigenschaften anzunehmen. Auch Bakterien nutzen eine Vielzahl von kleinen ncRNAs. Diese, auch als sRNAs bezeichnet, fungieren als Schlüsselregulatoren der Genexpression während der Adaption an eine sich rasch ändernde Umwelt oder während einer virulenten Phase. Die Mechanismen, wie diese sRNAs wirken – direkt durch einen Antisense-Mechanismus oder indirekt durch Sequestrierungsproteine –, werden zunehmend besser verstanden. Diagnostische and therapeutische Herangehensweisen: ncRNAs als Werkzeug und Ziel NcRNAs und besonders miRNAs sind ethiologisch für eine Vielzahl von Erkrankungen verantwortlich. Als Konsequenz hieraus werden sie immer häufiger als diagnostische oder therapeutische Werkzeuge eingesetzt, auch wenn dies bisher noch durch einige technische Einschränkungen limitiert wird. Viele miRNAs sind zahlreich und stabil in Körperflüssigkeiten vorhanden, z.B. Blut oder Urin, und könnten daher als diagnostische Marker dienen [13]. miRNAs im Blut könnten z.B. zur Feststellung einer Schwangerschaft herangezogen werden [13]. Da miRNAs bei Krebserkrankungen dereguliert sind, würde ihre Detektion im Blut zu einer exakten Diagnose führen. Die eindeutige Gewebespezifität der miRNAs kann sogar bei Metastasen mit unbekanntem Primärtumor zu einer präzisen Diagnose führen. Solche Diagnosen wurden erst mit Fortschritten in der Hochdurchsatz- Sequenzierungstechnologie möglich. Sie erlaubt es, in einem einzigen Durchlauf Millionen von Sequenzen zu erhalten. Man ist damit in der Lage, miRNAs geringster Konzentration nachzuweisen. Besonders bei Tumorerkrankungen konnte gezeigt werden, dass miRNAs eine Schlüsselrolle als Tumorsuppressor oder als Onkogen spielen. Daher könnten die Überexpression oder die Repression von tumorassoziierten miRNAs zu neuen Krebstherapien führen. Die Hauptprobleme, die sich für eine technische Anwendung von ncRNAs ergeben, umfassen die Verbesserung zuverlässiger Transfersysteme, Methoden zur Überwindung natürlicher Barrieren (z.B. Membrane), die effiziente Verteilung von ncRNA im Zytoplasma sowie ihre Stabilität und Spezifität. Die Einführung von Nukleinsäuren in einen Organismus kann auf verschiedene Weise erfolgen. Zum Beispiel können ncRNA-Moleküle mithilfe viraler Vektoren z.B. Adeno- oder Adenoassoziierte Viren (AAVs) oder Retroviren, in Zellen eingeschleust werden. RNA-Moleküle können auch direkt und ohne Expressionsvektoren verwendet werden. Kationische Liposomen oder Nanodrops fördern effizient die Aufnahme von Nukleinsäuren in Zellen, denn sie können mit der Zellmembran fusionieren. Bei Affen führte eine einzige Injektion spezifischer siRNA in Lipidpartikeln zu einer Akkumulation in der Leber und einem maximalen Silencing von >90 % der Ziel-mRNA. Ihre Wirkung kann bis zu 11 Tage danach beobachtet werden. Das Einschleusen der ncRNAs kann auch durch Kopplung des Moleküls an einen Antikörper oder einen Zelloberflächen- Marker erreicht werden, der das Eindringen in einen spezifischen Zelltyp erlaubt [14]. Normalerweise sind RNA-Moleküle empfindlich und werden durch RNasen gespalten, stabilere ncRNAs können aber durch chemische Modifikation gewonnen werden. Dies erreicht man durch Derivatisierung der 2'-OH-Gruppe des Riboseteils einzelner oder aller Nucleotide durch Methylierung (= Antagomire) [15] oder durch Verknüpfung mit der 4'-Methylengruppe eines Linkers (LNAs). Als Folge davon geht das C'-3-Atom der Ribose in die endo- Konformation über; gleichzeitig erhöht sich damit die Affinität und Spezifität. Mit Cholesterin an 2'-OH verknüpfte Antagomire oder LNAs wurden bei Mäusen und nicht menschlichen Primatenmodellen erfolgreich genutzt, um den Plasma-Cholesterinspiegel zu senken. Zielort ist dabei miR-122, eine miRNA, die am Cholesterinstoffwechsel der Leber beteiligt ist [15]. Ein entscheidender Vorteil einer solchen Vorgehensweise sind die im Vergleich zu den üblichen Medikamenten sehr geringen Nebenwirkungen. Schlussfolgerungen Nicht proteinkodierende-RNAs (ncRNAs) werden in vielen biologischen Reaktionswegen genutzt, ihre Beteiligung an einer Vielzahl von menschlichen Gesundheitsstörungen wurde ausführlich dokumentiert. Die Beteiligung derartiger ncRNA-Moleküle an molekularen Mechanismen, die zu Krankheiten führen, zeigt auch Wege zu neuen diagnostischen Verfahren und therapeutischen Anwendungen. Die Regulierung der ncRNAs erscheint schneller und effizienter zu sein als ihre direkte Wirkung auf Protein-kodierende Gene selbst. Es >90 % der Ziel-mRNA. Ihre Wirkung kann bis zu 11 Tage danach beobachtet werden. Das Einschleusen der ncRNAs kann auch durch Kopplung des Moleküls an einen Antikörper oder einen Zelloberflächen- Marker erreicht werden, der das Eindringen in einen spezifischen Zelltyp erlaubt [14]. Normalerweise sind RNA-Moleküle empfindlich und werden durch RNasen gespalten, stabilere ncRNAs können aber durch chemische Modifikation gewonnen werden. Dies erreicht man durch Derivatisierung der 2'-OH-Gruppe des Riboseteils einzelner oder aller Nucleotide durch Methylierung (= Antagomire) [15] oder durch Verknüpfung mit der 4'-Methylengruppe eines Linkers (LNAs). Als Folge davon geht das C'-3-Atom der Ribose in die endo- Konformation über; gleichzeitig erhöht sich damit die Affinität und Spezifität. Mit Cholesterin an 2'-OH verknüpfte Antagomire oder LNAs wurden bei Mäusen und nicht menschlichen Primatenmodellen erfolgreich genutzt, um den Plasma-Cholesterinspiegel zu senken. Zielort ist dabei miR-122, eine miRNA, die am Cholesterinstoffwechsel der Leber beteiligt ist [15]. Ein entscheidender Vorteil einer solchen Vorgehensweise sind die im Vergleich zu den üblichen Medikamenten sehr geringen Nebenwirkungen. Schlussfolgerungen Nicht proteinkodierende-RNAs (ncRNAs) werden in vielen biologischen Reaktionswegen genutzt, ihre Beteiligung an einer Vielzahl von menschlichen Gesundheitsstörungen wurde ausführlich dokumentiert. Die Beteiligung derartiger ncRNA-Moleküle an molekularen Mechanismen, die zu Krankheiten führen, zeigt auch Wege zu neuen diagnostischen Verfahren und therapeutischen Anwendungen. Die Regulierung der ncRNAs erscheint schneller und effizienter zu sein als ihre direkte Wirkung auf Protein-kodierende Gene selbst. Es besteht deshalb berechtigte Hoffnung, auch bisher „medikamentenresistente“ Proteine zu beeinflussen. Die Identifikation aller kleinen und größeren ncRNAs ist unabdingbar, um alle pathologischen Befunde vollständig zu verstehen und um neue therapeutische Ansätze bei Erkrankungen zu entwickeln.
Foto: Rederstorff M. und Hüttenhofer A., |
L&M 3 / 2010
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Die Autoren:Weitere Artikel online lesenNewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |