|
Computer-Aided Drug Design mit Moleküloberflächen
Computer-Aided Drug Design mit MoleküloberflächenWissenschaftler und WahrsagerWir kennen die Argumente – in silico-Wirkstoffdesign hilft, die Synthese auf die viel versprechendsten Verbindungen zu beschränken und spart Geld sowie Tierversuche. Kaum ein Antrag auf Fördergelder auf diesem Gebiet lässt die Tatsache unerwähnt, dass computer-aided drug design (CADD) ein fester Bestandteil der Entwicklung von new chemical entities (NCEs) sei. Fast alle F&E-Abteilungen bei großen Pharmaunternehmen beschäftigen eine große Anzahl von Computerchemikern. Wir liegen zu 95 % falsch
Objektiv gesehen ist die Situation weniger rosig. Fachleute wissen, dass zum Beispiel die Trefferquote in prospektives high-throughput virtual screening selten 5 % übersteigt. Anders ausgedrückt – wir liegen zu 95 % der Zeit falsch. Es gibt viele weitere Beispiele, die zeigen, dass heutige in silico-Techniken höchstens als semiquantitativ einzustufen sind. Warum? Die Antwort liegt in der Komplexität biologischer Systeme und im Rauschniveau sowohl von computerbasierten als auch experimentellen Techniken in der Wirkstof fentwicklung. Idealerweise sollte CADD genau so zuverlässig sein wie Crashsimulationen in der Autoindustrie, die als prädiktiv anzusehen sind und es uns wirklich ersparen, dass endlos Autos gegen harte Gegenstände gefahren werden müssen. Crashsimulationen basieren aber auf relativ einfachen physikalischen Prinzipien. Der Fluch der Vereinfachung
Schlechte Daten sind aber nicht das einzige Problem. CADD leidet unter seiner eigenen Geschichte. Die erste Ausgabe von Yvonne Martin’s Quantitative Drug Design: A Critical Evaluation [3] wurde vor drei Jahrzehnten veröffentlicht. Zu dieser Zeit dauerte eine MNDO-Optimierung der Ascorbinsäure auf einem „Mini-Supercomputer” vierzig Minuten (bei einem Listenpreis von 1,2 Millionen Dollar) im Vergleich zu weniger als einer Sekunde auf einem modernen Laptop. Das Problem der Wechselwirkungen
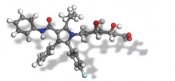
Die jetzige Situation ist ungewöhnlich. Das Problem der Berechnung der Wechselwirkungsenergie zwischen zwei Molekülen ist komplett verstanden – selbst wenn eins davon ein Makromolekül ist. Klassische Kraftfelder von hoher Qualität beherrschen solche Aufgaben problemlos. Moderne Kombinationen von Hard- und Software können biologische Systeme in relativ kurzen Zeiten berechnen. Dieses Wissen für biologische Systeme umzusetzen, ist aber aufgrund zweier Hauptprobleme sehr schwierig – Solvatation und Konformationssuche. Moleküle sind anders Ein Problem der heutigen Techniken ist, dass sie auf nichtphysikalischen Molekülmodellen basieren. Streng genommen existieren Atome und Bindungen nicht innerhalb von Molekülen. Kraftfelder funktionieren, weil die Eigenschaften von Atomen oder Gruppen innerhalb von Molekülen normalerweise transferierbar sind – aber auch weil wir frei sind, so viele verschiedene Atomtypen zu definieren, wie wir brauchen. Innerhalb der Born-Oppenheimer-Näherung sind „echte“ Moleküle ein Meer von nicht unterscheidbaren Elektronen im Feld der stationären, positiv geladenen Atomkerne. Chemiker können wenig mit dieser Beschreibung anfangen. Sie führt aber zu einer Sicht von Molekülen als unregelmäßigen flexiblen Festkörpern, die über die bekannten intermolekularen Kräfte (Pauli-Abstoßung, Coulomb-Wechselwirkungen, Dispersion und Ladungstransfer) mit ihrer Umgebung (einschließlich mit anderen Molekülen) wechselwirken. Durch diese veränderte Sichtweise mutiert das „ball and stick“-Modell (Abb. 1) in eine Darstellung der Moleküloberfläche. Das MEP (molekulare elektrostatische Potential), das Coulomb-Wechselwirkungen zwischen Molekülen beschreibt, stellt nur eine einer Reihe von lokalen Eigenschaften dar, die die verschiedenen intermolekularen Wechselwirkungen beschreiben [6]. Diese Eigenschaften können eingeingesetzt werden, um Funktionen zu konstruieren, die die Solvatation des Moleküls in Wasser, seine Hydrophobizität und seine intermolekularen Bindungseigenschaften beschreiben. Diese Funktionen können wiederum für Analyse- oder Visualisierungszwecke auf Moleküloberflächen projiziert werden. Das Ende vom Lied – Moleküle als Felder Molekulare Felder beinhalten noch mehr Information. Das elektrostatische Feld zum Beispiel wird als die erste Ableitung des MEP gerechnet. Da das Feld eine Vektoreigenschaft darstellt, beinhaltet es Richtungsinformation, die im MEP nicht enthalten ist. Die doppelseitige Abbildung zeigt das Beispiel der Überlagerung des elektrostatischen Feldes eines Wirkstoffmoleküls (gelbe Pfeile) und seines Rezeptors (grüne Pfeile) im Ligand-Rezeptor-Komplex. Die Komplementarität der Felder der zwei Komponenten kodiert Information über ihre Coulomb-Wechselwirkung. Gewöhnlich stellen Moleküloberflächen unhandliche mathematische Objekte dar, sodass neue Methoden vonnöten sind, um sie kompakt analytisch darzustellen. Eine solche Methode stellt das Anpassen der Oberfläche an eine Reihe von sphärischen Kugelfunktionen dar. Dies ermöglicht gleichzeitig sehr kompakte Speicherung der Oberfläche und schnelle analytische Handhabung im Computer mit der zusätzlichen Möglichkeit, die räumliche Auflösung der Oberfläche zu ändern [7]. Die ersten Schritte Richtung eines umfassenden oberflächenbasierten Modellierungssystems sind schon gemacht – es bleibt aber viel zu tun. tim.clark@chemie.uni-erlangen.de Abb. 1 „Balls and sticks" Darstellung einer möglichen Konformation von Lipitor. Literatur
[1] Modelling the Chemistry: time to break the mould? , T. Clark in EuroQSAR 2002: Designing drugs and crop protectants, M. Ford, D. Livingstone, J. Dearden and H. V. d. Waterbeemd (Eds) Blackwell Publishing, Oxford, 2003 , 111-121. Foto: © Molcad GmbH, Darmstadt |
L&M 5 / 2010
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Der Autor:Weitere Artikel online lesenNewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |