|
Forscher
>
Prof. Dr. Andreas Tauch
>
de.NBI und GFBio
de.NBI und GFBioBioinformatische Infrastrukturen zum Management und zur Analyse großer biologischer Datenmengen in den LebenswissenschaftenLeistungsfähige Forschungsinfrastrukturen entwickeln heutzutage eine herausragende Triebkraft für die lebenswissenschaftliche Forschung und ermöglichen es, komplexe und teils auch interdisziplinäre wissenschaftliche Fragestellungen zu adressieren. Beispiele für bioinformatische Infrastrukturprojekte in Deutschland sind das Deutsche Netzwerk für Bioinformatik-Infrastruktur (de.NBI) und die Deutsche Vereinigung für biologische Daten ( German Federation for Biological Data; GFBio). Das Big-Data-Problem in den Lebenswissenschaften
Die Lebenswissenschaften, also Biologie und Medizin, werden als die wissenschaftlichen Leitdisziplinen des 21. Jahrhunderts gesehen. Die Grundlage für diese Einschätzung ist eine Technikrevolution, durch die mithilfe neuester Technologien molekulare Vorgänge auf der zellulären Ebene in ihrer Gesamtheit beschrieben werden können. Zu diesen sogenannten Omics-Technologien zählen neben der Genomik auch die Transkriptomik, Proteomik und Metabolomik. Ein markantes Merkmal dieser neuen Technologien ist das Generieren riesiger Datenmengen in immer kürzer werdenden Zeiträumen. Die moderne medizinische Forschung zeichnet sich zudem durch den zunehmenden Einsatz datenintensiver Bildgebungsverfahren aus.
Deutsches Netzwerk für Bioinformatik-Infrastruktur (de.NBI)–www.denbi.de Der offizielle Start dieser vom Bundesministerium für Bildung und Forschung (BMBF) geförderten Infrastrukturinitiative erfolgte nach einer sechsmonatigen Konzipierungsphase im März 2015. Mit diesem fünfjährigen Förderprojekt verfolgt das BMBF das Ziel, die Verfügbarkeit von Rechen- und Speicherkapazitäten sowie von Datenressourcen und bioinformatischen Werkzeugen in den Lebenswissenschaften zu verbessern und nachhaltig sicherzustellen. Das Netzwerk besteht derzeit aus acht Leistungszentren (Tab. 1), in denen Kompetenzen thematisch gebündelt sind und deren Aktivitäten von einer übergeordneten Koordinierungsstruktur zentral gesteuert werden. Für die Koordinierung des Netzwerkes fiel die Wahl des BMBF auf Professor A. Pühler von der Universität Bielefeld. Die acht Leistungszentren mit insgesamt 22 nationalen Projektpartnern sind thematisch voneinander abgegrenzt und verfügen über spezifische Expertisen und Ressourcen in der Bioinformatik, die sie im Rahmen dieser Initiative dem wissenschaftlichen Nutzer als Serviceangebot zur Verfügung stellen. Durch die Bereitstellung von Programmen und Softwarelösungen sowie durch eine direkte wissenschaftliche Projektunterstützung durch das de.NBI-Servicepersonal ermöglicht das Netzwerk die bioinformatische Bearbeitung großer Datenmengen für experimentell arbeitende Wissenschaftler. Bei de.NBI handelt es sich somit um ein nationales Infrastruktur- und Servicenetzwerk. Daneben steht weiterhin das Training von wissenschaftlichem Personal im Vordergrund der Netzwerkaktivitäten. Dazu bieten die Leistungszentren eine Vielzahl von ein- und mehrtägigen Trainingskursen an, über deren Termine und Kursinhalte die Projektwebseite unter dem Link www.denbi.de informiert. Vervollständigt wird das Serviceprogramm durch Symposien, Workshops und einwöchige Sommerschulen, in denen neue Entwicklungen auf dem Bioinformatiksektor und auf dem Gebiet der Big-Data-Analyse beleuchtet werden. Deutsche Vereinigung für biologische Daten (GFBio) – www.gfbio.de Das GFBio Projekt startete im Dezember 2013 mit einer Konzipierungs- und Entwicklungsphase von 18 Monaten. Nach der erfolgreichen Evaluation des Projektes im Juni 2015 befindet sich GFBio jetzt in der zunächst auf drei Jahre veranschlagten Implementationsphase. GFBio wird von Dr. Michael Diepenbroek als Leiter von PANGAEA an der Universität Bremen koordiniert. Neben den Universitäten bilden sieben deutsche Museen- und Sammlungsarchive sowie ausgewählte molekularbiologische Archive und Dienste den Verbund (Tab. 2). Während der Konzept- und Entwicklungsphase konnten in einem integrativen Ansatz bereits die infrastrukturellen Ressourcen von 19 deutschen Schlüsselinstitutionen gebündelt sowie grundlegende Datenquellen und Dienste harmonisiert und über das GFBio Datenportal (www.gfbio.org) angeboten werden. Durch die breite internationale Vernetzung der beteiligten Partner ist eine Einbettung von GFBio in das internationale Umfeld und die dort genutzten Standards und maßgeblichen Datenmanagementstrategien gegeben. GFBio – das Rundum-sorglos-Paket für wissenschaftliches Datenmanagement
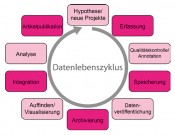
GFBio als Infrastruktur adressiert die wesentlichsten Anforderungen im Datenmanagement verschiedenster Interessengruppen und Disziplinen einschließlich der von Forschungsinstituten, einzelnen Wissenschaftlern, deutschen Naturkundemuseen, Entwicklern wissenschaftlicher Software sowie von großen Forschungsprojekten, -gruppen und -netzwerken. GFBio wird in der Lage sein, heterogene Daten aus verschiedensten Disziplinen zu integrieren und ermöglicht damit die effiziente Zusammenstellung und Nutzung von großskaligen und komplexen Datenprodukten für innovative Ansätze in der Biodiversitätsforschung. Dazu gehört neben Datenmobilisierungs-, Standardisierungsund Archivierungsaufgaben auch die Bereitstellung von Integrations-, Visualisierungs- und Analysewerkzeugen. Der GFBio Terminologie- Server ermöglicht den Einsatz neuer semantischer IT-Technologien. Dies wird es den Wissenschaftlern erleichtern, qualitativ hochwertige Daten aus den verschiedenen Archiven effektiv aufzufinden und zu nutzen. Die von GFBio angebotenen Dienste decken den kompletten Datenlebenszyklus von der Erfassung der Rohdaten bis zur Veröffentlichung wissenschaftlicher Artikel (siehe Abbildung) ab. Um das Bewusstsein für ein besseres Datenmanagement zu schärfen, rundet GFBio sein Profil durch eine breite Öffentlichkeitsarbeit, Kursangebote und einen Helpdesk ab.
Abb. Der von GFBio unterstutzte Zyklus von Daten in einem Forschungsprojekt – von der Idee (Hypothese) uber die Datensammlung und Qualitatssicherung und Datenpublikation bis zur Datenintegration und Analyse sowie der klassischen Publikation des Artikels in einer Fachzeitschrift.
-> tauch@cebitec.uni-bielefeld.de Bilder: © istockphoto.com| Eraxion, 4X-image, Vernon Wiley |
L&M 2 / 2016
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Die Autoren:Weitere Artikel online lesenNewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |