|
Forscher
>
Prof. Dr. H.P. Fiedler
>
Isolierung und taxonomische Charakterisierung von Actinomyceten – ein „Muss“ für ein erfolgreiches Wirkstoffscreening
Isolierung und taxonomische Charakterisierung von Actinomyceten – ein „Muss“ für ein erfolgreiches WirkstoffscreeningSuche nach neuen WirkstoffproduzentenActinomyceten sind neben den Pilzen unsere wichtigsten Antibiotikaproduzenten. Die Suche nach neuen Antibiotika ist nach über 60 Jahren der Antibiotikaforschung heute nur dann erfolgreich, wenn neue Spezies aus bislang unerforschten Quellen isoliert und im Wirkstoffscreening eingesetzt werden. Den Isoliermethoden und der taxonomischen Charakterisierung kommt daher ein besonderer Stellenwert zu. Actinomyceten kommen in allen Ökosystemen unserer Erde vor, sie können aus Böden, aus Meeressedimenten wie auch aus symbiontischen Lebensgemeinschaften, wie z.B. die pflanzliche Rhizosphäre und der Insektendarm, isoliert werden. Nachdem schätzungsweise über 10 Mio. Actinomycetenstämme in den vergangenen 60 Jahren weltweit in verschiedenen Wirkstoff- screenings auf Antibiotikaproduktion untersucht wurden, vorwiegend in Labors der chemisch-pharmazeutischen Industrie, stellt sich die Frage, weshalb wir heute noch Actinomyceten isolieren und taxonomisch charakterisieren sollen. Die Notwendigkeit, die Suche nach neuen Wirkstoffen wieder aufzugreifen und zu intensivieren, ist im fortschreitenden Auftreten resistenter, vor allem multiresistenter klinisch relevanter, pathogener Keime begründet, die nur noch mit wenigen Reserveantibiotika bzw. überhaupt nicht mehr therapierbar sind. Die grampositiven Actinomyceten sind unter den Bakterien und neben den Pilzen die bedeutendsten Wirkstofflieferanten der Natur. Nach Berechnungen von Richard Baltz [1] sind weltweit ca. 107 Actinomycetenisolate in Stammsammlungen konserviert. Diese, im ersten Augenblick als beachtlich erscheinende Menge ist jedoch verschwindend klein im Vergleich zum Vorkommen in der Natur und entspricht nur dem winzigen Bruchteil der Spitze eines Eisbergs. Ein Gramm Erdboden enthält 106–107 Actinomyceten, wobei die obersten 10cm der Erdschicht die größte Diversität an Mikroorganismen aufweist. Diese oberste Schicht entspricht einem Gewicht von ca. 1013t, die die unvorstellbare Anzahl an 1025–1026 Actinomyceten enthält [1]. Selbst wenn man davon ausgeht, dass nur 10% der in den Böden vorhandenen Mikroorganismen neue Spezies sind, ist deren verfügbare Menge astronomisch groß. Das zugängliche Reservoir zur Isolierung neuer Stämme ist daher unbegrenzt. Es umfasst nicht nur unsere terrestrischen Böden, sondern auch die Meeressedimente und die in der Rhizosphäre von Pflanzen und in der Darmflora von Insekten symbiontisch vergesellschafteten Mikroorganismen.
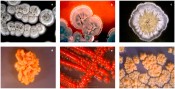
Abb.1 Kolonien von a) Streptomyces sp. Acta 3034; b) Streptomyces sp. AK 623; c) Streptomyces sp. Tü 6392; d) Verrucosispora maris AB-18-032; e) Rhodococcus sp. NTK 336; f) Tsukamurella pseudospumae Acta 1857
Erfolgversprechende Quellen für neue Mikroorganismen Zum Auffinden von neuen Wirkstoffproduzenten sind sowohl neue Screeningverfahren wie auch die Verwendung von qualitativ hochwertigem biologischen Material aus neuen, bislang unerforschten Quellen [2] erforderlich. Werden diese Voraussetzungen nicht berücksichtigt, so führt die Wirkstoffsuche stets zum Wiederentdecken von Produzenten altbekannter Antibiotika und Antitumorwirkstoffe. Solch unerforschte Quellen zur Isolierung von Actinomyceten findet man in mannigfaltigen Ökosystemen. Die Erschließung möglichst vieler dieser unerforschten Quellen erfordert aus meiner Erfahrung die internationale Zusammenarbeit mit taxonomisch ausgerichteten Mikrobiologengruppen. Unsere Arbeiten wurden vorwiegend in Zusammenarbeit mit Professor Michael Goodfellow von der University of Newcastle und Professor Alan Bull von der University of Kent durchgeführt. Wir haben Actinomyceten aus tropischen Böden des südamerikanischen, afrikanischen und asiatischen Kontinents isoliert, aus ariden Wüstenböden der Atacama Chiles und des australischen Outbacks, aus dem Permafrostboden der Antarktis wie auch aus Böden der gemäßigten Klimazonen, insbesondere aus der Rhizosphäre von Pflanzen (Kooperation mit Professor Rüdiger Hampp, Universität Tübingen) sowie Endosymbionten aus Insekten (Kooperation mit Professor Konrad Dettner, Universität Bayreuth) [3].
Abb.2 Ausstrich einer 1:100 verdünnten Bodenprobe auf Huminsäure-Vitamin-Agar als selektives Isoliermedium für Actinomyceten
Marine Habitate, vor allem Meeressedimente, sind ebenfalls eine erfolgversprechende Quelle zur Isolierung neuer Actinomyceten [4], sie sind jedoch wesentlich schwieriger zu beschaffen, nur mit hohen Kosten zugänglich, und sie erfordern zwingend eine internationale Kooperation mit Meeresorganisationen. Aus einem Sediment der Sea of Japan wurde mit Verrucosispora maris die erste marine Spezies der Gattung Verrucosispora isoliert [5], aus der wir Hemmstoffe der para-Aminobenzoesäure- und Folsäurebiosynthese, das Abyssomicin C und atrop-Abyssomycin C, isolieren konnten [3]. Aufgrund ihres neuen Wirkortes hemmen die Abyssomicine multiresistente wie auch vancomycinresistente Staphylokokken. Eine weitere neue marine Spezies der Gattung Verrucosispora, V. fiedleri, wurde aus einem Sediment des Raune Fjords, Norwegen isoliert [6] und als Produzent der Proximicine beschrieben, einer Gruppe neuer antitumoraler Aminofuran-Antibiotika [3]. Aus Sedimenten der atlantischen und der pazifischen Tiefseegräben konnten ebenfalls zahlreiche neue Spezies der Gattungen Streptomyces, Micromonospora, Dermacoccus und Williamsia isoliert [7] und als potente Produzenten neuer antibakterieller und antitumoraler Wirkstoffe beschrieben werden. Zu erwähnen sind u.a. (i) das Albidopyron, ein Enzymhemmstoff aus einer neuen Streptomycetenspezies, die aus einem Sediment im Nordatlantik isoliert wurde; (ii) das Caboxamycin, ein antibakterieller und antitumoraler Wirkstoff aus einer neuen Streptomycetenspezies, die aus einer Sedimentprobe des Kanarischen Beckens aus einer Tiefe von 3.814m isoliert wurde; (iii) aus der gleichen Sedimentprobe wurde eine weitere neue Streptomycetenspezies isoliert, die den Enzymhemmstoff Benzoxacystol produziert; (iv) die Dermacozine, antioxidativ wirkende Substanzen aus Dermacoccus abyssi, der aus einem Sediment des Marianengrabens aus einer Tiefe von 10.898m isoliert wurde [3]. Aus all diesen Quellen konnten neue Spezies und neue Gattungen der Bakterienordnung Actinomycetales isoliert werden. Innerhalb dieser Ordnung waren es erwartungsgemäß Spezies der Gattung Streptomyces, die zahlenmäßig am häufigsten vorkamen. Streptomyceten sind bekanntermaßen die artenreichste Gruppe der Actinomyceten. In Abbildung 1 ist eine kleine Auswahl der isolierten Actinomyceten dargestellt. Selektive Isolierung von Mikroorganismen Der selektiven Isolierung von Actinomyceten kommt ein besonders hoher Stellenwert zu, sofern neue Wirkstoffproduzenten im Fokus stehen. Nonomura & Hayakawa [8] inkubierten die Bodenprobe in einer Lösung aus 6% Hefeextrakt und 0,05% SDS, um bei 40°C ruhende Actinomycetensporen zu aktivieren. Diese Vorbehandlung führt zu einer signifikanten Erhöhung der CFUs (colony forming unit). 1:100 verdünnte Lösungen werden anschließend auf Agarplatten mit Selektivmedien ausgestrichen und bis zur Koloniebildung bzw. Sporulation bei 27°C inkubiert. Als besonders erfolgreiche Isoliermedien für Actinomyceten haben sich Huminsäure-Vitamin-Agar (Abb.2), Raffinose-Histidin-Agar und Stärke-Casein-Nitrat-Agar erwiesen. In jedem Falle ist der Zusatz von Nystatin (50µg/ml) erforderlich, um das Wachstum von Pilzen zu unterdrücken. Die sichtbaren Kolonien können nun unter dem Binokular selektioniert und auf ein klassisches Agarmedium ohne Antibiotikumzusatz transferiert werden, wie z.B. auf ISP-2-Agar. Eine Übersicht der Methoden findet sich bei Bergey’s Manual of Systematic Bacteriology [9]. Dereplikation der Isolate Nach der erfolgreichen Isolierung von Reinkolonien steht die Dereplikation der Isolate an, um zum einen Dubletten zu eliminieren und zum anderen die Gattungs- und Spezieszugehörigkeit zu ermitteln. Eine gängige Methode ist die Kombination der Zugehörigkeit zu „color-groups“ [10] mit der Vollsequenzierung des 16S rRNA-Gens und Vergleich der Sequenzen mit denen der nächsten Verwandten in GenBank. Sofern eine entsprechende Gerätetechnik zur Verfügung steht, ist die Pyrolyse-Massenspektrometrie (PyMS) eine leistungsfähige Ganzzellmethode zur Charakterisierung von Mikroorganismen. Die Probenmenge beträgt nur 100µg und die Analysenzeit pro Probe 90s. Durch Automatisierung können 100 Proben pro Zyklus analysiert werden. Eine zuverlässige Zuordnung zu Gattung und Spezies erfordert zwingend die Ermittlung der morphologischen und chemotaxonomischen Merkmale, wie Farbe des Substrat- und Luftmyzels, Sporenfarbe und Sporenform, Bestimmung der Art der Diaminopimelinsäure und taxonomisch relevanter Zucker im Peptidoglykan sowie das Fettsäure- und Menachinonmuster [11]. Das Wirkstoffscreening Aus den vorangegangenen Erörterungen wird ersichtlich, dass ein erheblicher Aufwand betrieben werden muss, um neue Actinomycetengattungen und -spezies isolieren zu können, um sie anschließend in diverse Screeningmethoden einzuschleusen mit dem Ziel, neue Wirkstoffe zur Therapie von Infektionskrankheiten und tumoralen Erkrankungen aufzufinden. Als eine erfolgreiche Screeningmethode hat sich das HPLC-Diodenarray-Screening erwiesen [3,12], die es ermöglicht, in Kulturfiltraten und Rohextrakten das Vorkommen von neuartigen Sekundärstoffen zu ermitteln und gleichzeitig bekannte Substanzen zu identifizieren. In den vergangenen Jahren wurde diese Methode durch die Onlinekopplung mit ESI-MS perfektioniert, und sie ermöglicht mit hoher Sicherheit eine Prognose über das Vorliegen neuartiger Sekundärstoffe. Aufgrund der Analysenzeit pro Probe (ca. 20min) und der individuellen, nicht automatisierbaren Auswertung eignet sich die Methode nicht für den Probenhochdurchsatz, sondern für den von uns favorisierten Weg, dem Screening mit selektiv isolierten, dereplizierten Mikroorganismen aus ausgewählten und unerforschten Habitaten. Ein nicht zu verschweigender Nachteil dieser Screeningmethode besteht darin, dass zur Erstellung einer HPLC-DAD-MS-Datenbank eine umfangreiche Naturstoffsammlung für Referenzzwecke zur Verfügung steht, und dass die Elutionsparameter der Substanzen von der mobilen und stationären Phase und von der verwendeten Gerätetechnik abhängig sind. Eine Standardisierung ist zwingend erforderlich.
Literatur Bild: © istockphoto.com| pixalot |
L&M 10 / 2014
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Der Autor:Weitere Artikel online lesenNewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |