|
Forscher
>
Prof. Dr. Thomas Carell
>
Erweiterung des genetischen Systems
Erweiterung des genetischen SystemsCode erkanntDer genetische Code codiert alle Informationen, die in jeder Zelle für die korrekte Funktion und Interaktion der Zelle mit der Umgebung notwendig sind. Aufgebaut wird er aus vier unterschiedlichen Molekülen, den so genannten kanonischen Watson-Crick-Basen Adenin, Cytosin, Guanin und Thymin. Die Abfolge dieser vier Basen im DNA-Doppelstrang, abgekürzt A, G, C und T, stellt den genetischen Code dar. Seit der Entdeckung der DNA-Doppelhelixstruktur durch Watson und Crick im Jahr 1953 wissen wir, wie diese vier Basen sich im DNA-Molekül anordnen. Seit dieser Zeit beschäftigt sich ein großer Teil der internationalen Wissenschaftsgemeinschaft mit der Frage, wie die Abfolge der vier Basen durch Leseproteine erkannt und in Proteine, d. h. in die Funktionseinheiten der Zelle, übersetzt wird. Seit Langem ist bekannt, dass es neben diesen vier Standardbasen noch eine weitere Base gibt, das so genannte 5-Methylcytosin (Abb.1). Diese Base wird genutzt, um Gene an- und abzuschalten. Da alle Zellen das gleiche DNA-Molekül und damit die gleiche Sequenzinformation als Hardware besitzen, sich unterschiedliche Zelltypen aber bezüglich ihrer Funktion deutlich unterscheiden (man vergleiche nur eine Nervenzelle mit einer Hautzelle), bedarf es einer übergeordneten Informationsebene jenseits der Sequenzinformation. Auf dieser Ebene wird festgelegt, welche zelltypspezifischen Gene aktiv sind und welche der entsprechende Zelltyp stillgelegt hat. Diese Informationsebene wird vom Forschungsgebiet der Epigenetik untersucht. Bis zum Jahr 2009 galt es als erwiesen, dass unser genetisches System auf diesen 4 + 1-Basen aufgebaut ist.
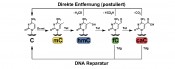
Abb.1 Darstellung der neuen DNA-Basen hmC, fC und caC sowie der derzeit postulierten Reparatur/Entfernungsprozesse. 5-Hydroxymethylcytosin Im Jahr 2009 wurde entdeckt, dass eine weitere Base in unserem Genom eine große Rolle spielen könnte, die heute als sechste Base des Genoms bezeichnet wird. Es handelt sich um die Base 5-Hydroxymethylcytosin (hmC, Abb.1) [1]. Bislang kannte man diese Base als oxidativen Schaden, d.h., es wurde angenommen, dass 5-Hydroxymethylcytosin ein Abbauprodukt ist, das, wenn es im Genom auftritt, schnell durch Reparaturprozesse entfernt wird. Solche DNA-Abbauprodukte gibt es viele. Sie entstehen durch reaktive Sauerstoffspezies, die in unseren Mitochondrien im Rahmen der Zellatmung freigesetzt werden. Diese Sauerstoffspezies greifen die DNA-Basen an und überführen sie in so genannte oxidative Schäden. Diese Schäden, zu denen bislang auch das 5-Hydroxymethylcytosin gezählt wurde, werden durch Reparaturenzyme im Genom effizient aufgespürt und beseitigt. Im Jahr 2009 wurde bekannt, dass Hydroxymethylcytosin nicht nur ein oxidativer Schaden ist, sondern dass es aktiv in unserem Genom produziert wird. Verantwortlich hierfür sind spezielle Enzyme, die man Tet Enzyme nennt und von denen bislang drei (Tet1 bis Tet3) entdeckt wurden [2]. Bei diesen Tet-Enzymen handelt es sich um Oxidationsenzyme, die mithilfe des Kofaktors Ketoglutarat und mit einem im aktiven Zentrum gebundenen Eisenatom Oxidationsprozesse auslösen. Diese Enzyme oxidieren die fünfte Base des Genoms 5-Methylcytosin selektiv in einem ersten Schritt zu 5-Hydroxymethylcytosin. Diese Entdeckung hat das gesamte Gebiet der Epigenetik, d.h. das Wissenschaftsgebiet, das sich mit der Frage beschäftigt, wie Gene an- und abgeschaltet werden, zentral verändert. Bedenkt man, dass das regulierte An- und Abschalten von Genen die Basis der Zelldifferenzierung ist, so begreift man, dass die Entdeckung von Hydroxymethylcytosin auch die Stammzellforschung derzeit massiv beeinflusst. Pluripotente Stammzellen, die nach der Befruchtung der Eizelle durch die Samenzelle entstehen, sind die Basis für die Entwicklung aller Gewebe. Während des Entwicklungsprozesses müssen die entsprechenden Genabschnitte, die zu dem spezifischen Gewebe führen, selektiv aktiviert, andere selektiv abgeschaltet werden. Das Abschalten und Anschalten von Genen sowie die Prozesse, die diesen Schaltprozessen zu Grunde liegen, sind die Basis für die Entwicklung eines komplexen Organismus aus einer befruchteten Eizelle, einer so genannten Zygote. Es wird nun vermutet, dass die gezielte Oxidation von Methylcytosin zu Hydroxymethylcytosin, also von der fünften zur sechsten Base des Genoms, für diese An- und Abschaltprozesse von besonderer Bedeutung sind. Tatsächlich zeigen Messungen, dass Hydroxymethylcytosin gerade in embryonalen Stammzellen in überraschend hohen Konzentrationen vorliegt, was diese Theorie derzeit stützt [2]. Komplexe Oxidationsprozesse Weitere Forschungen lieferten Hinweise, dass die Oxidationsprozesse von Methylcytosin zu Hydroxymethylcytosin wesentlich komplexer sind. So wurden im Jahr 2011 zwei weitere vom Cytosin abgeleitete DNA-Basen gefunden, die man heute als siebente und achte Base des Genoms bezeichnen kann [3–5]. Es handelt sich dabei um Formyl- und Carboxycytosin (fC und caC, Abb.1), d. h., um die höheren Oxidationsprodukte des Hydroxymethylcytosins. Es konnte gezeigt werden, dass die Tet-Enzyme nicht nur Methylcytosin zu Hydroxymethylcytosin oxidieren, sondern auch weitere Oxidationsschritte eben zum besagten Formyl- und Carboxycytosin durchführen (Abb.1). Wer diese Oxidationsprozesse steuert und warum diese sukzessiven Oxidationsprozesse von Bedeutung sind, ist bis heute völlig unklar. Darüber hinaus verstehen wir nicht, in welchem Maße diese neuen Basen Hydroxymethyl-, Formyl- und Carboxycytosin eigene biochemische Funktionen haben, d.h. spezifische Proteine rekrutieren, die anschließend an den An- und Abschaltprozessen der Gene beteiligt sind. Mithilfe massenspektrometrischer Verfahren wird derzeit weltweit nach Proteinen gesucht, die hochspezifisch diese neuen Basen binden, um Einblicke zu gewinnen, welche biochemischen Prozesse von diesen neuen DNA-Basen kontrolliert werden [6]. Dabei ergibt sich immer deutlicher das Bild, dass diese Oxidationsprozesse an der DNA und hier speziell an den Cytosinbasen maßgeblich für das kontrollierte An- und Abschalten von Genen sind. So konnte u.a. gezeigt werden, dass Hydroxymethylcytosin von der humanen DNA-Reparaturmaschinerie nicht erkannt wird. Es verbleibt also im Genom, obwohl Reparaturprozesse eine Großzahl modifizierter Basen, die im Laufe eines Tages in jeder Zelle entstehen, erkennen und herausschneiden. Die Tatsache, dass Hydroxymethylcytosin im Genom verbleibt, deutet bereits an, dass es möglicherweise eine viel größere Bedeutung hat, als wir bisher erahnen können. Im Gegensatz zu Hydroxymethylcytosin werden die Formyl- und Carboxylverbindungen sehr wohl von Reparaturprozessen aus dem Genom herausgeschnitten, was uns vermuten lässt, dass diese Basen erzeugt werden, damit die Natur mC gezielt aus dem Genom entfernen kann. Neben der Reparatur von hmC, fC und caC kommen hierbei auch einfache direkte „Entfernungen“ d.h. Umwandlungen in C infrage, basierend auf einer Dehydroxymethylierung von hmC, Deformylierung von fC oder Decarboxylierung von caC. Was wir heute nicht verstehen, ist, ob Reparaturprozesse oder diese direkten Umwandlungen tatsächlich mit der Reaktivierung von Genen zusammenhängen oder ob doch nur Basen entfernt werden, die durch ungewünschte Überoxidation im Genom entstehen. Sind Formyl- und Carboxycytosin Schäden, die durch eine zu große fehlerhafte Aktivität der Tet-Oxidasen gebildet werden? Oder ist die Bildung von Formyl- und Carboxylcytosin ein gewollter, biochemisch notwendiger Schritt, der Prozesse auslöst, die wir bisher noch nicht verstehen? Kürzlich konnte auch gezeigt werden, dass die Tet-Enzyme auch die kanonische Base Thymin in kleinen Mengen zu Hydroxymethyluracil aufoxidieren können. Hydroxymethyluracil ist auch eine Base, die durch die humane DNA-Reparatur effizient erkannt und repariert wird. Damit stellt sich auch in diesem Fall die Frage, ob Hydroxymethyluracil eine Base ist, die durch eine möglicherweise fehlerhafte Oxidation entsteht oder ob die Oxidation und nachfolgende Reparatur biochemisch unbekannte Prozesse auslösen? Alle diese Beobachtungen legen nahe, dass das An- und Abschalten von Genen intensiv mit den unterschiedlichen DNA-Reparaturprozessen verknüpft ist. Genabschnitte werden methyliert und dadurch stillgelegt. Durch Oxidationsprozesse am Methylcytosin entstehen Hydroxymethylcytosin, Formylcytosin und Carboxylcytosin, die im Fall des Hydroxymethylcytosins stabil im Genom verbleiben. Im Fall von Formylcytosin und Carboxylcytosin werden demgegenüber DNA-Reparaturprozesse oder direkte Umwandlungen zu C ausgelöst. Ein ähnliches Bild ergibt sich für Thymin, das durch die gleichen Tet-Enzyme zu Hydroxymethyluracil aufoxidiert wird, welches ebenfalls Reparaturprozesse auslöst. Diese Reparatur- bzw. Umwandlungsprozesse führen in allen Fällen zum Ersatz der hochoxidierten Basen hmC, fC, caC und auch hmU durch die kanonischen Basen Cytosin (C) und Thymin (T), womit der Prozess aufs Neue starten kann. So entsteht ein Kreislauf aus Methylierung (nur im Fall der C-Basen), Oxidation und Entfernung der aufoxidierten Basen, gefolgt vom Ersatz durch „frisches“ Cytosin bzw. Thymidin. Es ergibt sich das Bild eines dynamischen Genoms, in dem die Sequenzinformation statisch vorliegt, aber die Aktivität der Gene durch Oxidation und Reparatur geregelt wird. Welche exakten biochemischen Prozesse die oxidierten Basen auslösen, liegt noch im Dunkeln. Aber was immer die neuen vier Basen auch biochemisch steuern mögen, eines steht heute schon fest: Unser genetisches System ist viel komplizierter als angenommen.
Literatur
Foto: © www.de.123 |
L&M 8 / 2013
Das komplette Heft zum kostenlosen Download finden Sie hier: zum Download Der Autor:Weitere Artikel online lesenNewsSchnell und einfach die passende Trennsäule findenMit dem HPLC-Säulenkonfigurator unter www.analytics-shop.com können Sie stets die passende Säule für jedes Trennproblem finden. Dank innovativer Filtermöglichkeiten können Sie in Sekundenschnelle nach gewünschtem Durchmesser, Länge, Porengröße, Säulenbezeichnung u.v.m. selektieren. So erhalten Sie aus über 70.000 verschiedenen HPLC-Säulen das passende Ergebnis für Ihre Anwendung und können zwischen allen gängigen Herstellern wie Agilent, Waters, ThermoScientific, Merck, Sigma-Aldrich, Chiral, Macherey-Nagel u.v.a. wählen. Ergänzend stehen Ihnen die HPLC-Experten von Altmann Analytik beratend zur Seite – testen Sie jetzt den kostenlosen HPLC-Säulenkonfigurator!© Text und Bild: Altmann Analytik ZEISS stellt neue Stereomikroskope vorAufnahme, Dokumentation und Teilen von Ergebnissen mit ZEISS Stemi 305 und ZEISS Stemi 508ZEISS stellt zwei neue kompakte Greenough-Stereomikroskope für Ausbildung, Laborroutine und industrielle Inspektion vor: ZEISS Stemi 305 und ZEISS Stemi 508. Anwender sehen ihre Proben farbig, dreidimensional, kontrastreich sowie frei von Verzerrungen oder Farbsäumen. © Text und Bild: Carl Zeiss Microscopy GmbH |